科学家可以使用液体活检样本来检测和表征血液中存在的各种癌症生物标志物。健康人和未患癌症的人体内没有这些生物标志物。1 因美纳为癌症生物标志物研究提供创新的新一代测序(NGS)液体活检解决方案,以分析:
- 循环肿瘤细胞(CTC)
- 游离DNA(cfDNA)和循环肿瘤DNA(ctDNA)
- 游离RNA(cfRNA)和循环肿瘤RNA(ctRNA)
- 用于检测和分析的循环蛋白
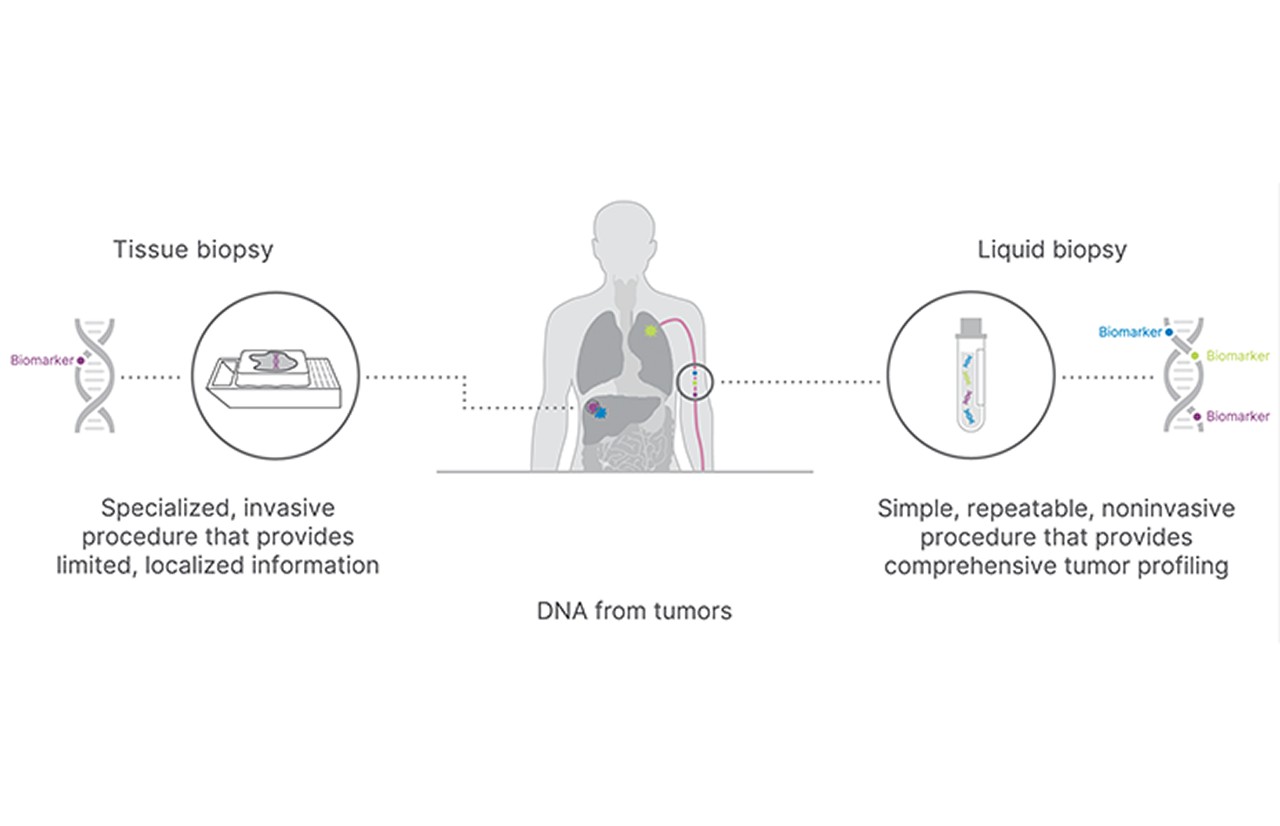
将样本可及性与可靠分析相结合,用于癌症研究
科学家可以使用液体活检样本来检测和表征血液中存在的各种癌症生物标志物。健康人和未患癌症的人体内没有这些生物标志物。1 因美纳为癌症生物标志物研究提供创新的新一代测序(NGS)液体活检解决方案,以分析:


这本20余页的电子手册汇总了已发表的、全面的实验流程,帮助您借助NGS和芯片技术对液体活检样本进行深度表征。内容涵盖循环肿瘤细胞(CTC)测序、循环肿瘤DNA(ctDNA)甲基化分析、ctDNA测序以及无细胞RNA(cfRNA)测序。
Your email address is never shared with third parties.
一些挑战限制了组织活检在转化和临床癌症研究中的应用,包括:2
CTC是从肿瘤脱落到血液中的细胞。多项研究表明,CTC具有转移潜力,并且与各种癌症类型的侵袭性或晚期疾病以及不良预后有关。虽然CTC可以作为基于液体活检的癌症表征的生物标志物,但它们在血液中含量非常少(平均7.5 mL血液中仅< 1至< 50个CTC),这对检测和表征而言是一个重大挑战。基于NGS的技术可以高通量检测低水平的CTC,为癌症筛查和早期检测开辟了一个令人兴奋的前沿领域。
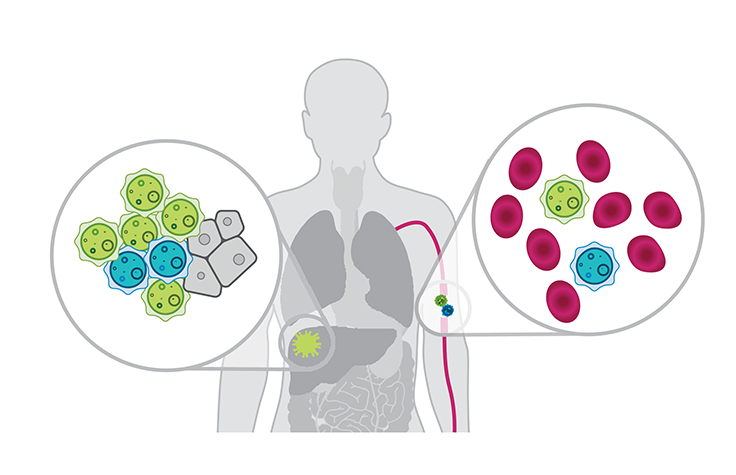
单细胞分离和单细胞测序方法的技术进步使研究人员能够在基因组、转录组和表观基因组水平上对单个CTC进行详细分析。将液体活检与CTC的单细胞测序相结合可以阐明导致肿瘤生物学的细胞异质性。10
异常的甲基化模式是许多癌症的标志。DNA甲基化的变化发生在肿瘤发生的早期,会导致大多数基因组处于低甲基化状态,而CpG岛处于高甲基化状态。
这种整体甲基化模式的变化导致基因组不稳定和肿瘤抑制基因沉默,从而推动肿瘤进展和转移。重要的是,多项研究表明,ctDNA的甲基化模式再现了原发细胞/组织中存在的模式。这表明ctDNA甲基化的液体活检分析可用于癌症的早期检测、原发组织的分析、微小残留病(MRD)的监测、治疗反应的监测等。11,12

血液中的多种细胞类型以及癌细胞可以释放cfRNA。这些ctRNA是潜在的生物标志物,可以通过液体活检进行检测,以识别特定癌症、检测癌症的发生、揭示原发组织、阐明疾病的分子机制、监测治疗反应等。13
与在每个细胞/组织中都相同的DNA(遗传变异除外)不同,RNA在不同细胞类型和组织之间呈现动态的差异表达。这使得ctRNA可用于检测癌症并有望用于在体内定位癌症。RNA表达的多样性还有助于利用ctRNA在疾病早期确定癌症亚型并对其进行分类,这一点很重要,因为即使是与同一器官或组织相关的癌症,其进展、治疗方案和预后也存在很大差异。14

了解 NovaSeq X 在使用 TruSight Oncology ctDNA v2 检测试剂盒处理 ctDNA 样本时与 NovaSeq 6000 的性能对比。结果显示,使用 NovaSeq X 系列时,能够在显著缩短运行时间的同时,保持相同的高水平性能表现。
作为基因组学技术的先锋,因美纳提供经过验证的集成工作流程和创新解决方案,适用于癌症研究的液体活检分析物测序。
有兴趣了解更多关于液体活检在癌症研究中的应用吗?
Your email address is never shared with third parties.