Abstract
- 针对急性髓系白血病、骨髓增生异常综合征、骨髓瘤和慢性淋巴细胞白血病等癌症的研究,已显著推动了个性化治疗的进步。
- 传统检测手段通常包括核型分析、FISH(荧光原位杂交)、染色体芯片及基因panel。
- 而全基因组测序仅需单一流程,即可检出与这些疾病相关的所有关键异常和变异类型。
简介
1973年,芝加哥大学的Janet Rowley发现了慢性骨髓性白血病(CML)的细胞遗传学基础——在她研究的几乎每一个细胞中,都发现了9号染色体和22号染色体之间的易位。这一发现建立在1959年Peter Nowell和David Hungerford对CML患者费城染色体的鉴定之上,这标志着首次发现与癌症的直接遗传联系。20世纪90年代,分子表征发现,这种相互易位事件会产生一个融合基因——BCR-ABL1,它来自B细胞受体BCR和ABL1的激酶结构域。BCR-ABL1融合基因的鉴定为靶向治疗铺平了道路。伊马替尼(商品名Gleevec)是首个针对CML中BCR-ABL1蛋白开发的酪氨酸激酶抑制剂。该药物于2001年获得美国食品和药物管理局批准,标志着癌症治疗进入了一个新纪元,在CML患者中取得了前所未见的成功,五年生存率高达89%。
大多数CML病例(超过95%)都具有典型的t(9;22)易位特征。其他髓系恶性肿瘤,如急性骨髓性白血病(AML)和骨髓增生异常综合征(MDS),以及淋巴系恶性肿瘤,如多发性骨髓瘤(MM)和慢性淋巴细胞白血病(CLL),都具有遗传异质性的特点,变异种类繁多。例如,在AML中,主要的遗传学发现包括反复发生的染色体易位,如t(8;21)、inv(16)和t(15;17),以及FLT3、NPM1、CEBPA、IDH1/2和DNMT3A等基因的突变,这些发现定义了具有不同预后影响的特定AML亚型。这些基因改变会影响疾病行为、治疗反应和风险分级。这种遗传结果的多样性是其他骨髓性和淋巴性血癌的典型特征。
白血病治疗的一般工作流程包括通过血细胞计数和骨髓活检进行初步诊断、分类、使用基因组工具进行风险分级,以及通过化疗、干细胞移植和维持疗法进行治疗。适当的分类和风险分级对患者管理至关重要,对干细胞移植和其他干预措施的关键临床决策也是必不可少的。
目前,血液癌症的基因和分子检测采用多模式方法,这些实验室结果被用于根据世界卫生组织和其他临床指南对疾病进行分类和风险分级。传统的细胞遗传学(核型分析)用于检测大的染色体变化,而荧光原位杂交(FISH)则有助于确定特定的重排和隐性易位。聚合酶链反应(PCR)和反转录PCR可检测特定基因突变(如FLT3、NPM1)和融合转录本,有助于诊断和监测最小残留病(MRD)。新一代测序(包括靶向panel)为风险分级提供了全面的突变分析。染色体芯片和光学基因组图谱方法可在特定情况下使用。
事实证明,全基因组测序(WGS)是一种高效的替代方法,它能提供全面的基因组图谱,检测所有相关的基因异常,包括染色体变化、结构变异以及细胞遗传学和FISH等传统方法可能遗漏的隐性突变。由此实现了更有效的分类和风险分级,一项针对AML/MDS的研究表明,与目前的标准检测方法相比,使用WGS时,近25%的患者获得额外的诊断结果,约17%的患者获得不同的风险分级1。WGS还能帮助新兴领域(如发现基于血浆的MRD启用途径的靶点)摆脱使用骨髓穿刺液进行纵向检测的做法。WGS通过在统一的工作流程下将多项检测合并为一项检测,简化了诊断流程,周转时间更短,仅需五天左右。虽然WGS在历史上一直被认为是一种昂贵的检测方法,但测序成本的降低使其变得更加经济可行。此外,该方法已被纳入美国国立综合癌症网络AML/MDS管理指南,并享受医疗保险报销。
工作原理
现有的因美纳产品和工作流程可用于从血液恶性肿瘤患者的标本中生成全面、高质量的基因组。根据研究情况,从外周血和/或骨髓穿刺样本中提取的DNA适合作为起始材料。对于体细胞变异的检测,建议采用更深的平均覆盖度,因为关键事件可能只出现在部分细胞中。为了达到更高的测序深度,通常使用相对较少的文库进行混合。因此,在选择样本标签时应小心谨慎,避免由于接头序列相似而造成数据丢失。请参阅我们的《标签序列接头混合指南》,或联系您当地的因美纳支持团队了解更多信息。
| 类别 | 名称 | 说明 |
|---|---|---|
| 文库制备 | Illumina DNA PCR-Free Prep Illumina DNA Prep |
标准WGS文库制备 |
| 测序仪和流动槽 | 配备S2/S4的NovaSeq™ 6000 配备10B/25B的NovaSeq™ X和NovaSeq™ X Plus |
大容量基因组测序 |
| 生物信息学 | 带血红素方案的DRAGEN v4.3 Somatic |
用于血红素WGS研究的定位和变异检出调整 |
| 解读和报告 | Illumina Connected Insights | 肿瘤学应用的三级分析平台 |
表1:可增强血液恶性肿瘤WGS研究能力的现有因美纳产品
概述
对血液恶性肿瘤进行全面的基因组分析需要评估多种变异类型,包括小变异(单核苷酸变异和插入缺失小于50 bp)、拷贝数改变(CNA)、结构变异(SV)(如易位)和杂合性缺失(LoH)计算。DRAGEN Somatic WGS Heme Tumor Only方案结合使用变异检出、覆盖深度分析、断裂端检测和其他算法,可为血液恶性肿瘤相关变异提供准确而全面的结果。
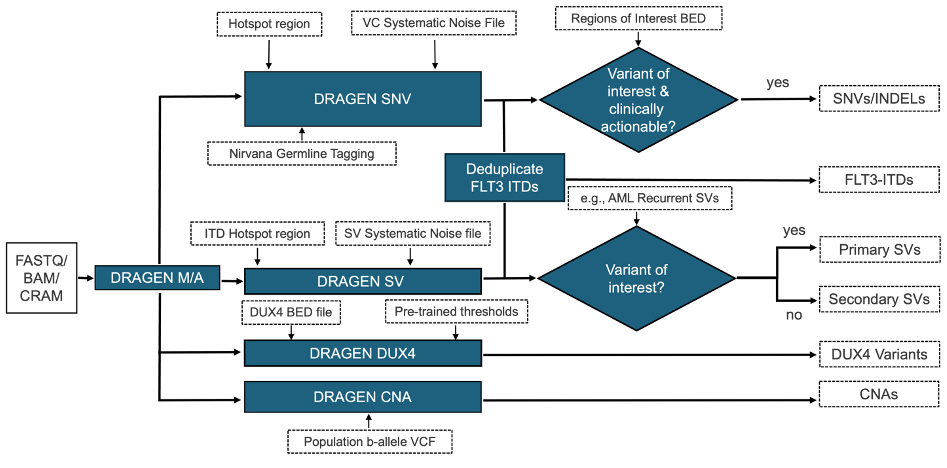
| 分析灵敏度(检测限) | |
|---|---|
| 平均覆盖度 | 140× |
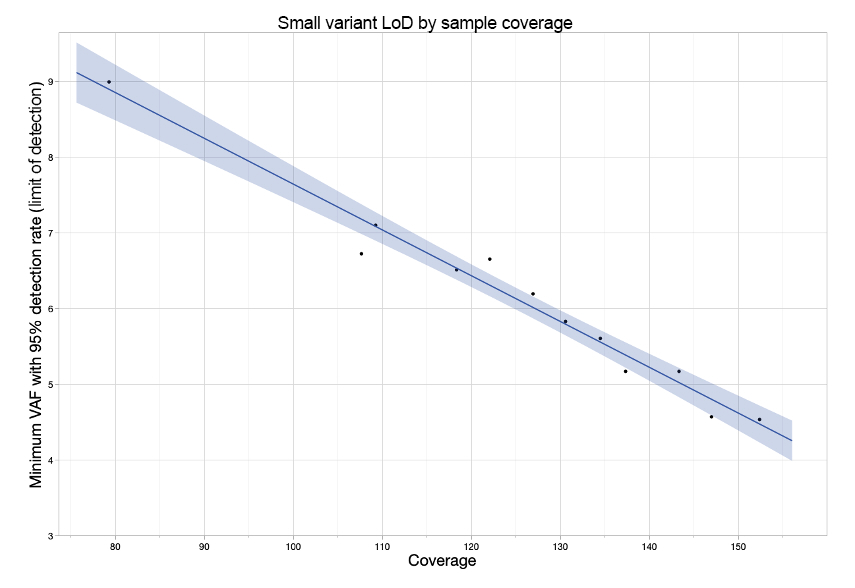
| 小变异(VAF) | 0.05 |
| 结构变异LoD(VAF) | 0.07 |
| 0.5 Mb–5 Mb CNA缺失(倍数变化) | 0.09 |
| 0.5 Mb–5 Mb CNA重复(倍数变化) | 1.09 |
| 5 Mb-10 Mb CNA重复(倍数变化) | 1.07 |
| 0.5 Mb–5 Mb LoH(肿瘤纯度) | 0.17 |
| 5 Mb-10 Mb LoH(肿瘤纯度) | 0.15 |
表2:140×覆盖度下确定的小变异、结构变异、CNA和LoH的分析灵敏度(检测限)
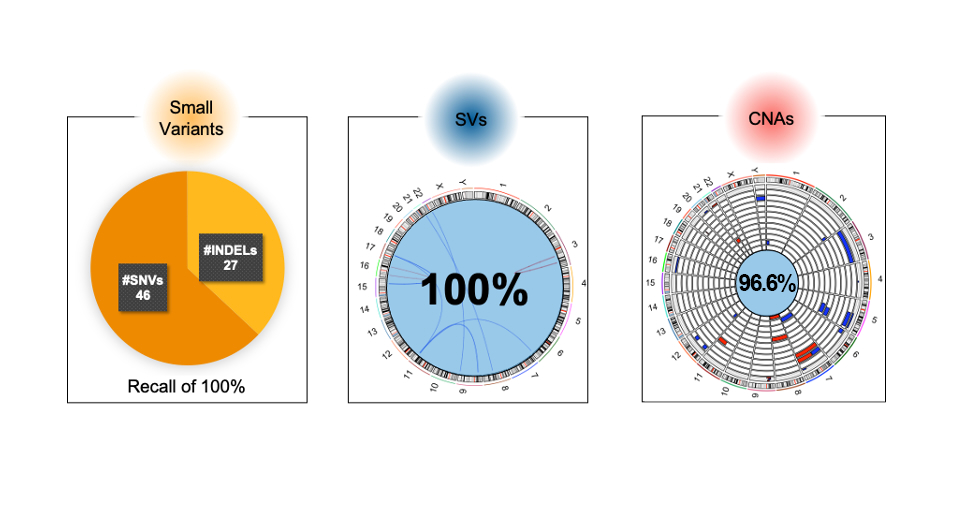
图3:WGS对血液临床样本的变异检测性能——DRAGEN在一组23个血液恶性肿瘤患者的临床样本中准确检测出73个小变异(31个基因)、10个结构变异(SV,7个基因)以及29个拷贝数变异(CNA)中的28个。
我们与研究人员合作,对23个患有血液恶性肿瘤的临床样本进行了PCR-Free WGS,平均覆盖度约为220×(见图3)。我们的团队在这组样本中正确检测出所有临床相关的小变异(n=73,包括4个FLT3-ITD)、结构变异以及超过96%的CNA。具体来说,共评估了两个长度分别约为45 Mb和56 Mb的倒位、8个易位、22个从4.5 Mb到100 Mb的CNA和7个全染色体事件。
为了进一步评估WGS的性能,我们评估了另外30份AML研究参与者的外周血或骨髓穿刺液标本,并将WGS的变异检出结果与TruSight Oncology 500的结果进行了比较。表3汇总了此项研究的结果,并显示了相关变异类型的高性能。
| AML样本的分析性能 | |
|---|---|
| AML样本数量 | 53 |
| 平均覆盖度 | ~200× |
| 小变异(SNV/插入缺失)准确度 | > 99% |
| 结构变异/拷贝数变异准确性 | > 95% |
| 分析特异性 | > 99.9% |
表3:研究队列中约200×覆盖度的WGS检测性能
基因组时代
发表在《血液》(Blood)杂志上的一篇论文指出:“在髓系肿瘤患者中,全基因组测序有可能取代传统的细胞遗传学和测序方法,提供快速、准确的全景变异分析”2。
与传统方法相比,因美纳针对血液恶性肿瘤的全面全基因组测序和信息学解决方案可在单一平台上实现统一的工作流程,从而提高实验室效率和临床疗效。从大型染色体重排到单核苷酸变异,每一个细节都可以通过这一新技术平台进行准确评估。如需更多支持,请联系您当地的客户经理或销售代表。
相关资源
illumina.com/products/by-type/sequencing-kits/library-prep-kits/illumina-dna-prep.html
illumina.com/products/by-type/sequencing-kits/library-prep-kits/dna-pcr-free-prep.html
help.dragen.illumina.com/product-guides/dragen-v4.3/dragen-recipes/somatic-wgs-heme-to
For more information or a DRAGEN trial license for academic use, please contact dragen-info@illumina.com.
参考文献
- Duncavage EJ, Schroeder MC, O’Laughlin M, et al. Genome Sequencing as an Alternative to Cytogenetic Analysis in Myeloid Cancers. N Engl J Med. 2021;384(10):924-935. doi:10.1056/NEJMoa2024534
- Duncavage EJ, Bagg A, Hasserjian RP, et al. Genomic profiling for clinical decision making in myeloid neoplasms and acute leukemia. Blood. 2022;140(21):2228-2247. doi:10.1182/blood.2022015853