简介
抗菌药物耐药性(AMR)是全球面临的主要威胁,对人类健康和经济发展具有重大影响1,2。想要了解全球和地方范围内AMR的发生和传播情况,需要准确、标准化的AMR监测数据。近年来,全球AMR的监测工作有所加强。2022年,全球126个国家、地区和州加入了世界卫生组织(WHO)全球抗微生物药物耐药性和使用监测系统(GLASS)2。然而,迄今为止收集的大部分AMR数据均来自临床环境中的个体样本,且通常仅限于可培养的致病物种3-6。想要在个体层面进行基因组AMR监测需要实验室具备极高的能力,这也是追踪AMR传播情况的阻碍之一4-6。
通过对废水等环境样本进行基因组筛查来监测AMR趋势,是个体样本检测的替代方法,可减轻公共卫生实验室样本采集的负担,并通过标准化分析方法提供社区层面的AMR频率和丰度信息5,6 。凭借这些优势,基于新一代测序(NGS)的废水AMR监测正在迅速获得关注,并有助于开发缓解全球AMR威胁的策略1。但是,尽管克服了个体采样中存在的一些挑战,但针对从环境来源收集的样本,基于NGS的AMR检测方法仍然面临挑战6。其中的一个挑战是AMR序列只占环境样本中总DNA的一小部分6。
鸟枪法宏基因组测序方法十分适用于优先考虑AMR标志物发现的应用或需要无假设微生物检测的应用。然而,用于环境AMR监测的鸟枪法需要深度测序,且可能无法在废水等复杂基质中提供足够的灵敏度7靶向NGS代表了一种独特、精确的环境AMR监测方法7。通过探针捕获开展靶向NGS,可以显著增加AMR靶标的reads,从而在降低测序深度的同时提供更高的AMR标志物检测灵敏度7。
泌尿道病原体/耐药基因检测试剂盒(UPIP,因美纳,仅供研究使用)专用于广泛和准确地检测微生物和AMR序列。与基于鸟枪法的采样方法相比,能够采用较低的测序深度检测与人类健康相关的序列。UPIP能够靶向170多种泌尿生殖系统病原体和3700多种AMR标志物,其中包括3600多种完整AMR基因,包含了综合抗菌药物耐药性数据库(CARD,版本3.2.5)8中超过75%的AMR基因,涵盖了美国疾病预防控制中心(CDC)9重点列表中75%的AMR威胁以及WHO10重点列表中83%的相关基因 (表1)。凭借广泛的靶标范围,UPIP AMR panel可作为主要候选产品用于开展基于NGS的环境样本AMR监测。为了检验UPIP试剂盒在废水AMR监测方面的性能,我们使用NovaSeq 6000系统在两种条件下(使用和不使用UPIP试剂盒)对废水样本进行了测序,每个样本平均生成300M条总reads,并比较了不同下采样测序深度的AMR产量 (表2)。
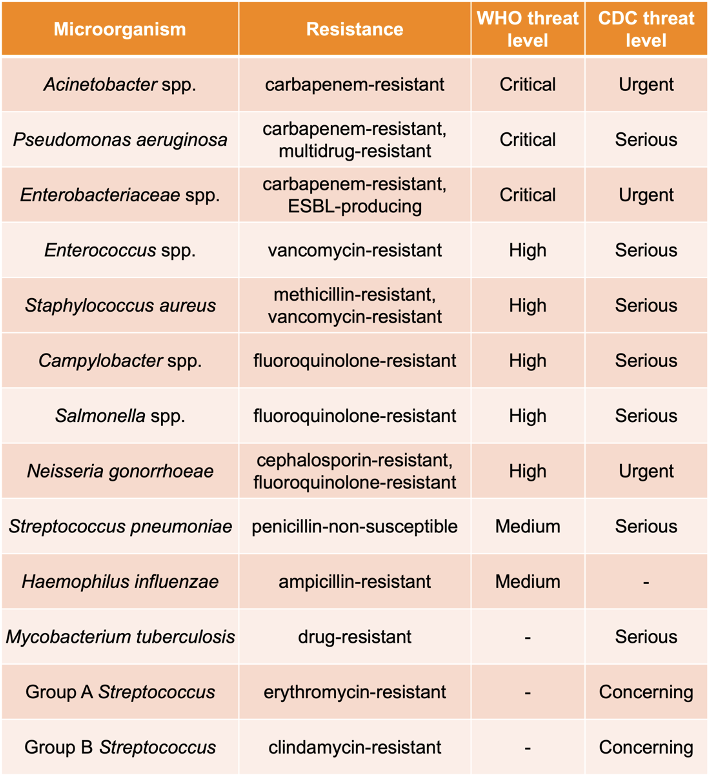
表1.UPIP靶向的CDC和WHO重点病原体。
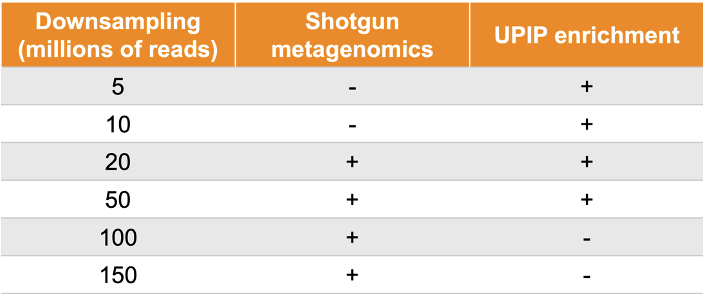
表2.样本测序平均reads数为300M。UPIP富集样本被下采样至5M、10M、20M和50M条reads。鸟枪法样本被下采样至20M、50M、100M和150M条reads。
UPIP富集显著提高了AMR检测效率:reads数量提高了>2500倍,测序深度提高了>3500倍,检测量提高了>3倍
从短读长测序数据中检测AMR标志物需要reads与AMR基因参考序列之间具有高比对覆盖度和一致性,这取决于从样本中生成的AMR reads产量、高质量且全面的参考数据库分析和软件解决方案。UPIP捕获探针旨在提高靶向AMR序列的测序产量,Explify UPIP数据分析BaseSpace Sequence Hub(BSSH)中提供的配套应用程序旨在生成具有高覆盖度和高一致性的共有序列和比对,从而准确检测AMR标志物。
我们使用Explify应用程序(v1.0.0)的样本组成功能估计了AMR序列(在使用及未使用UPIP检测的条件下)的测序reads比例。Explify样本组成功能使用基于kmer的方法将reads分为各种类别,例如“人类”、“细菌”或“AMR”。UPIP检测样本中CARD(版本3.2.5)AMR序列的reads中值为50%,而鸟枪法样本的reads中值为0.02%。即使测序深度降低30倍,通过UPIP检测,AMR类别的序列reads比例也高出2500倍 (图1)。平均而言,UPIP检测样本5M条总reads中有2.4M条属于AMR序列,而鸟枪法样本150M条总reads中有0.03M条属于AMR序列。总之,UPIP检测产生了更多分属于AMR序列的reads,能够支持下游检测算法。
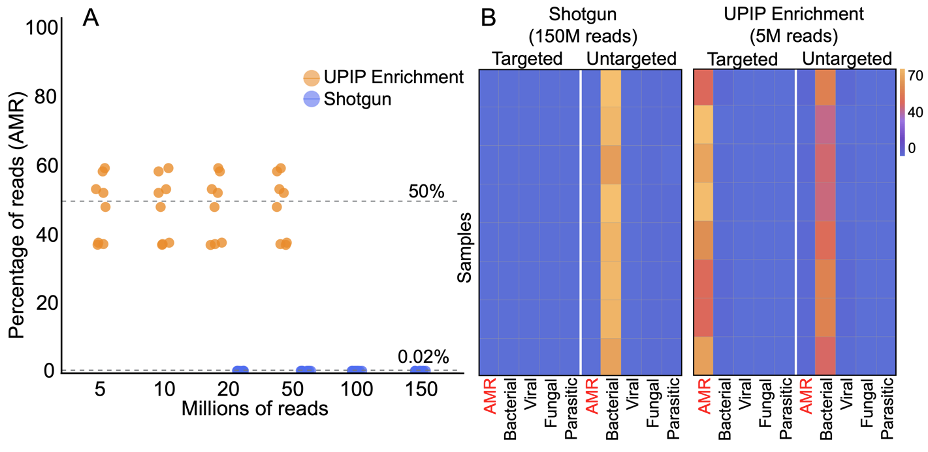
图1A:UPIP检测样本与鸟枪法样本中AMR标志物的reads百分比。虚线表示reads占比的中位数。
图1B:使用基于kmer的reads分类时,UPIP富集样本(总reads:5M)与鸟枪法样本(总reads:150M)的样本组成。左图显示的是UPIP靶向序列的总样本reads的百分比。右图显示的是非UPIP靶向序列的总样本reads的百分比。CARD(版本3.2.5)中75%的AMR基因是UPIP的靶标。
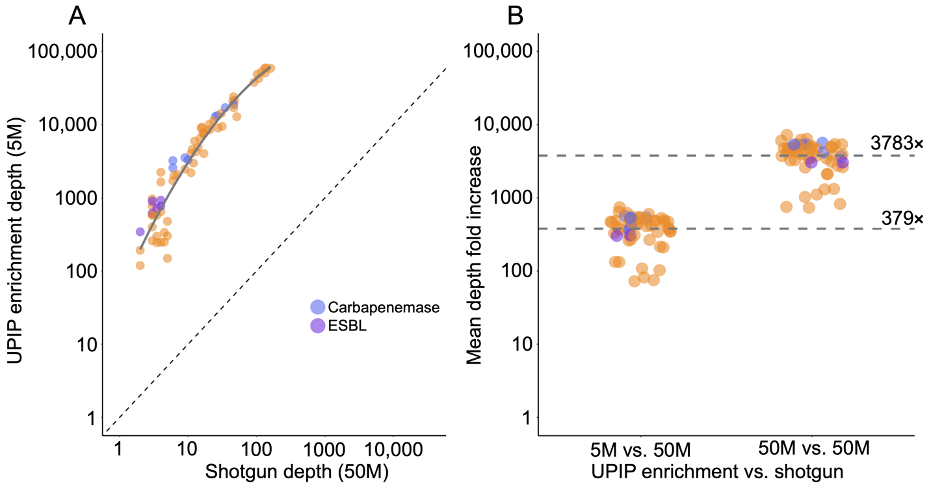
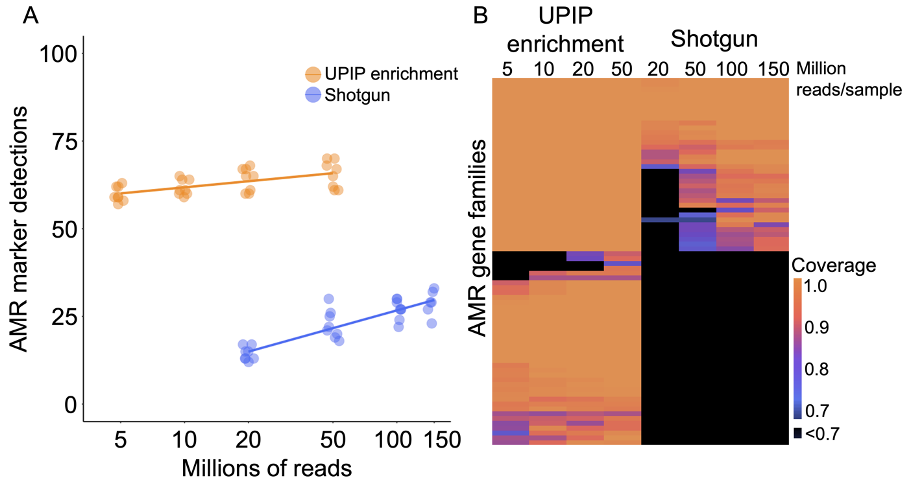
图3A:UPIP样本与鸟枪法样本中的AMR标志物检测。
图3B:在UPIP样本和鸟枪法样本中,比对覆盖度占序列长度的比例。
UPIP检测法提高了AMR变异检出能力
提高AMR测序效率对于准确检测低丰度AMR标志物和AMR标志物变异至关重要。因此,我们研究了UPIP检测法对AMR标志物变异检测的影响。Explify应用程序在所有UPIP样本(5M至50M条reads)中检测到了glpT基因中的E448K突变11。对于鸟枪法样本(20M至150M条reads),均未检测到相同的突变,但在reads数超过200M的8个原始鸟枪法样本中,有两个检测到了相同的突变。我们观察到,在UPIP样本中,该位置的平均深度为每百万总reads占85条reads,而鸟枪法样本中的读取深度为0.02,这表明,UPIP检测法在较低总测序深度下能够更好地检出废水样本中与耐药性相关的变异 (图4)。
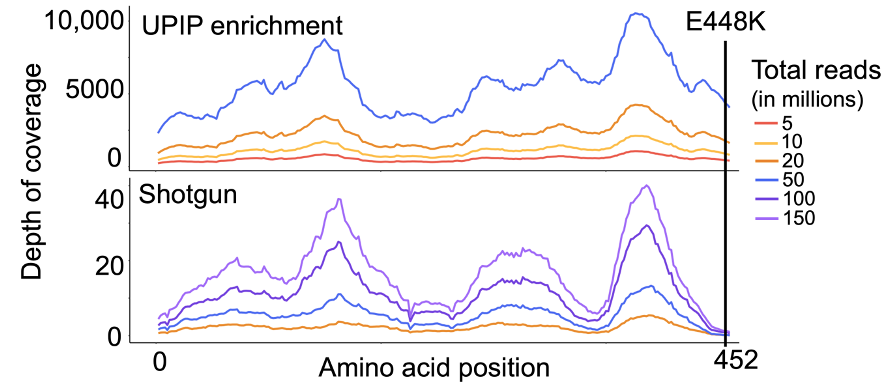
图4.UPIP样本与鸟枪法样本中glpT的平均覆盖深度。
检测重点病原体和潜在的AMR
UPIP靶向的170多种泌尿生殖系统病原体中报告了CDC9观察列表中的12种病原体(表1)。我们利用Explify应用程序的自动化微生物和AMR标志物检测结果,检查了与CDC重点威胁相关的病原体和AMR标志物的共同检测情况。我们发现,UPIP靶向的12种病原体中,有6种至少在一份样本中能够使用潜在的AMR标志物检测出来(图5)。在UPIP富集样本中(总reads至少为10M),均能检测到这12种病原体。在鸟枪法文库中检测到了肠球菌属(Enterococcus)不动杆菌属(Acinetobacter)和铜绿假单胞菌(Pseudomonas aeruginosa),后两种仅在总reads至少为100M的文库中检测到。在UPIP样本中,所有测序深度均检测到潜在的AMR标志物。值得注意的是,仅在UPIP富集样本中检测到抗万古霉素(例如vanA、vanB、vanC、vanD、vanRA、vanHA、vanYA)和抗甲氧西林(例如mecA、mecR1、mecI)的AMR标志物。在本分析中,潜在的AMR标志物是指与给定病原体具有已知关联的标志物或标志物组合。
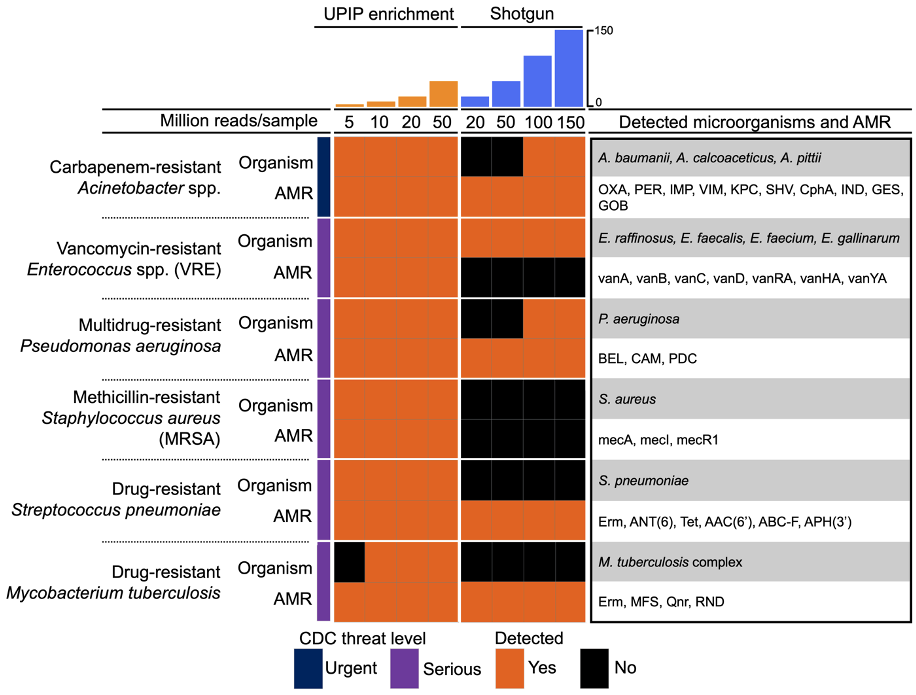
图5.CDC重点病原体和潜在AMR标志物的共同检测9。橙色表示使用该栏所示的测序深度,在至少一个样本中检测到病原体或潜在的AMR标志物。
结论
使用UPIP探针富集的废水样本,可以使靶向AMR标志物序列的reads数量增加 > 2500倍,靶向AMR标志物序列的reads深度增加 > 3500倍,每个样本的AMR标志物检测量增加 > 3倍,并且能够更可靠地同时检测CDC重点病原体和潜在AMR标志物。显著提高AMR检测效率可支持开展更广泛、更准确的AMR标志物检测,特别是废水等复杂样本类型中的低丰度AMR标志物和AMR标志物变异,而成本则大大低于传统鸟枪法宏基因组学测序的总成本。这项研究证实了精确宏基因组学(PMG)适用于优先考虑灵敏度和特异性的应用,并且UPIP检测法与Explify UPIP数据分析应用程序相结合为环境AMR监测提供了高效的分析解决方案。
如何将UPIP纳入我的AMR监测工作流程中?
UPIP富集试剂盒可支持多达384个标签,可在9小时内完成,手动操作时间为2小时,并且与所有因美纳仪器兼容12。点击此处,购买该试剂盒:https://www.illumina.com/products/by-type/sequencing-kits/library-prep-kits/urinary-pathogen-id-amr-enrichment-kit.html
Explify UPIP数据分析应用程序可通过BaseSpace Sequence Hub获取,可为UPIP富集试剂盒提供专用的基于云的信息学解决方案,具有灵活的分析选项、用户友好的界面和多种便用的报告格式。点击此处,获取更多信息,包括免费下载的演示项目:https://www.illumina.com/products/by-type/informatics-products/basespace-sequence-hub/apps/explify-upip-data-analysis.html
了解有关废水监测的更多信息,请单击: https://www.illumina.com/areas-of-interest/microbiology/public-health-surveillance/wastewater-surveillance.html
如有疑问,请访问idbd_biofx_explify_support@illumina.com,联系UPIP技术团队。.
参考文献:
1. Hendriksen RS, Munk P, Njage P, et al. Global monitoring of antimicrobial resistance based on metagenomics analyses of urban sewage. Nature Communications. 2019;10(1). doi: 10.1038/s41467-019-08853-3
2. Global antimicrobial resistance and use surveillance system (GLASS) report 2022. Geneva: World Health Organization; 2022. License: CC BY-NC-SA 3.0 IGO.
3. World Health Organization. Antimicrobial Resistance: Global Report on Surveillance. Geneva: World Health Organization; 2014.
4. Zignol M, Dean AS, et al. Twenty Years of Global Surveillance of Antituberculosis-Drug Resistance. N Engl J Med. 2016;375(1081-1089) doi:10.1056/NEJMsr1512438
5. Pruden A, Vikesland JP, Davis BC, et al. Seizing the moment: now is the time for integrated global surveillance of antimicrobial resistance in wastewater environments. Current Opinion in Microbiology. 2021;64. doi: 10.1016/j.mib.2021.09.013
6. Aarestrup FM, Woolhouse ME. Using sewage for surveillance of antimicrobial resistance. Science. 2020;367. doi:10.1126/science.aba3432
7. Guitor AK, Raphenya AR, et al. Capturing the Resistome: a targeted capture method to reveal antibiotic resistance determinants in metagenomes. Antimicrobial Agents and Chemotherapy. 2019;64. doi:10.1128/aac.01324-19
8. Alcock BP, Huynh W, Chalil R, et al. 2023. CARD 2023: expanded curation, support for machine learning, and resistome prediction at the Comprehensive Antibiotic Resistance Database. Nucleic Acids Research, 51, D690-D699.
9. CDC. Antibiotic Resistance Threats in the United States, 2019. Atlanta, GA: U.S. Department of Health and Human Services, CDC; 2019.
10. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. Geneva: World Health Organization; 2017(WHO/EMP/IAU/2017.12). Licence: CC BY-NC-SA 3.0 IGO.
11. Takahata S, Ida T, Hiraishi T, Sakakibara S, Maebashi K, Terada S, Muratani T, Matsumoto T, Nakahama C, Tomono K. Molecular mechanisms of fosfomycin resistance in clinical isolates of Escherichia coli. Int J Antimicrob Agents. 2010 Apr;35(4):333-7. doi: 10.1016/j.ijantimicag.2009.11.011. Epub 2010 Jan 13. PMID: 20071153.